安罗替尼作为一种新型多靶点的酪氨酸激酶抑制剂(TKI),可以通过肿瘤血管基质重编程、肿瘤细胞重编程和肿瘤免疫微环境重编程三大机制来杀伤肿瘤细胞。
下面就让我们一起走近安罗替尼抗肿瘤的第一大机制——肿瘤血管基质重编程,其余两大机制将在之后为大家详细叙述。
肿瘤血管生成是肿瘤生长和转移的关键
我们身体所需要的各种养分都需要通过血管来运输。相对于正常细胞而言,癌细胞对于营养物质的需求更高,细胞分裂需要更多的养分,转移入侵也需要养分,因此肿瘤在发生、发展的过程中会形成大量的新生血管,这些新生的血管为肿瘤的生长提供其所需要的营养供给,同时向远处扩散肿瘤细胞,在体内不同部位形成新的转移灶。
1945年Algire首次提出“肿瘤血管生成”的概念,1971年Folkman进一步提出血管生成与肿瘤生长、转移关系密切,并可以通过抑制血管生成,切断营养来源,达到抑制肿瘤生长的目的,也就是著名的“肿瘤饥饿疗法”。该理论的出现为抗肿瘤血管生成药物提供了研究方向和理论基础[1]。
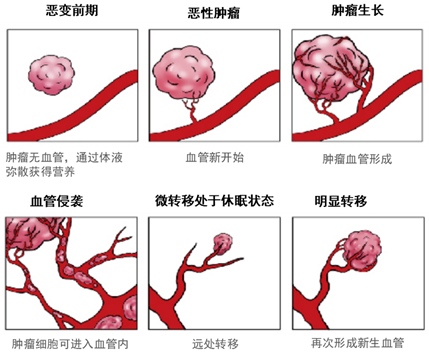
肿瘤血管新生
那么抗肿瘤血管生成药物究竟是如何发挥作用的呢?首先来了解一下肿瘤血管生成的机制
肿瘤血管生成是指在原有血管基础上生成以毛细血管为主的血管系统,并在肿瘤组织内建立血液循环的过程。肿瘤血管生成受多种促血管生成因子和血管生成抑制因子的调节[2]。
肿瘤组织的血管杂乱无章,并且存在大量的分支渗漏血管,血管通透性大大增加。这造成了间质流体压力升高以及血液灌注和氧合减少,导致肿瘤组织区域极度缺氧和药物输送不足,就像是漏水的管道,虽然源源不断地往身体输送着药物,但最后到达病灶内的却所剩无几。这些改变反过来又会引起肿瘤进展、侵犯周围组织、远处转移和化疗难治性[3]。
安罗替尼的肿瘤血管基质重编程作用,除了抑制血管新生,切断肿瘤营养来源之外,还能促进肿瘤血管正常化。增强药物递送,并导致渗漏减少,灌注增强,以阻止肿瘤的进一步恶化。正常化的肿瘤血管系统可以使化疗、靶向药物、免疫检查点抑制剂等的递送得到高度增强,更好地发挥抗肿瘤作用[3,4]。这就像把刚刚漏水的管道修理好,因此输送进身体的药物可以正常而且足量地到达病灶内,充分发挥作用。
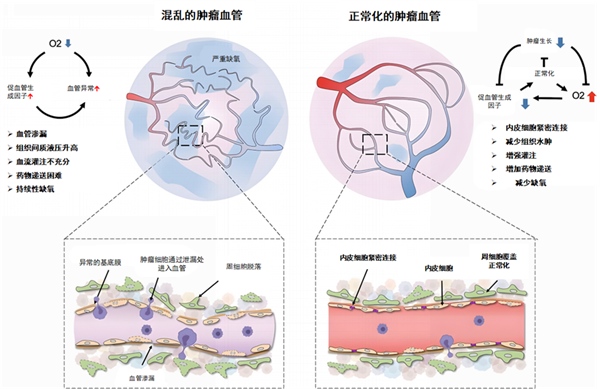
肿瘤血管正常化的示意图
安罗替尼作为我国自主研发的多靶点抗血管药物,是通过哪些方法达到抑制肿瘤血管生成,促进血管正常化的抗肿瘤效果呢?
釜底抽薪——安罗替尼有效抑制肿瘤血管新生,切断肿瘤营养来源
VEGF、PDGF和 FGF是三种主要的促血管生成因子,它们所介导的三条信号通路是调控肿瘤血管新生的主要机制[5]。
分子机制研究及动物实验显示,安罗替尼可同时抑制三种主要的促血管生成因子的受体,VEGFR、PDGFR和 FGFR,通过抑制血管形成相关的三条信号通路,全面阻断肿瘤血管新生,切断肿瘤营养来源,并且安罗替尼的抑制作用优于舒尼替尼和索拉非尼等其他药物[6-10]。
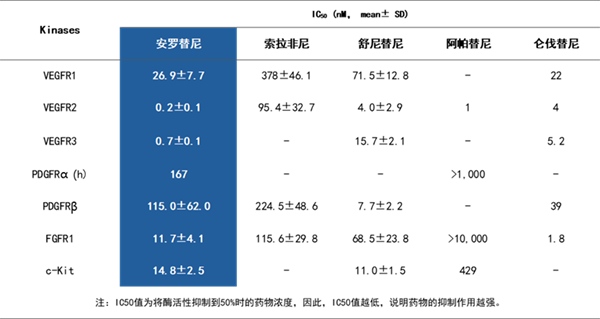
安罗替尼通过多通路阻断肿瘤血管新生
暗度陈仓——安罗替尼促进肿瘤血管正常化,增加药物递送
一项探讨安罗替尼对肿瘤细胞影响的动物实验显示,安罗替尼不仅可以显著抑制肿瘤新生血管,还可以改变肿瘤血管基质,促进血管正常化,从而改善血管形态和肿瘤血液灌注,使得血管渗漏减少,缺氧缓解,促进肿瘤间质液压恢复正常,进而增加化疗药物在肿瘤组织分布,进一步加强抗肿瘤效果[11]。
并肩作战——安罗替尼联合EGFR-TKI和化疗等,具有协同增效作用
研究显示,安罗替尼可增加化疗、靶向药物或免疫检查点抑制剂在肿瘤组织中的分布,是联合其他药物具有协同增效作用的机制之一。
在肺癌[12-16]、软组织肉瘤[17,18]、食管磷癌[19,20]、卵巢癌[21,22]等肿瘤治疗中,均有多项研究证实,安罗替尼联合化疗或靶向药物(如吉非替尼、尼拉帕利)可以显著控制肿瘤生长,改善患者生存。
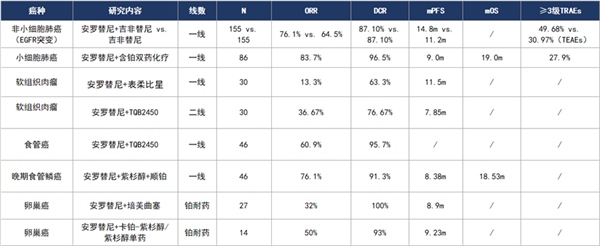
总结:
肿瘤血管生成与肿瘤的生长转移密切相关,成熟的肿瘤血管基质结构混乱导致组织极度缺氧,而且药物输送不足;安罗替尼作为多靶点小分子抗血管生成药物,可以通过有效抑制VEGF、PDGF、FGF信号通路,来抑制肿瘤血管新生;同时促进肿瘤血管的正常化,增加其他抗肿瘤药物递送至肿瘤组织,因此联合化疗、靶向治疗、免疫检查点抑制剂,具有协同增效的作用。
参考文献
[1] 鲁美钰,仲维兰,司春枫等.肿瘤血管生成机制及抗肿瘤血管新生的靶向药物研究进展[J].安徽医药,2018,22(05):798-802.
[2]林国享,朱小东.肿瘤血管生成的促进因素及其在肿瘤发生发展中的作用研究进展[J].广西医学,2020,42(03):334-337+346.
[3]Hellmut G Augustin, et al. Cancer Res. 2022 Jan 1;82(1):15-17.
[4]Jin-Sung Park, et al. Cancer Cell. 2016 Dec 12;30(6):953-967.
[5]姜婧琦,宋雨童,卢育彤等.抗血管生成靶向药物在肿瘤治疗中的研究进展[J].山东医药,2022,62(22):86-90.
[6]Xie C, et al. Cancer Sci. 2018;109(4):1207–1219.
[7]Lin B, et al. Gene. 2018 May 15;654:77-86.
[8]Unpublished data from Millipore.
[9]Wilhelm SM, et al. Cancer Res. 2004:64;7099-7109.
[10]Sun, Q. et al. Cancer Biol Ther. 2014;15(12):1635-45.
[11]Su, et al. Clin Cancer Res. 2022 Feb 15;28(4):793-809.
[12]Li Zhang, et al. 2022 ESMO Asia, LBA10.
[13]Tianqing Chu et al. Transl Lung Cancer Res. 2022 Jul;11(7):1394-1404.
[14]Wei Zhang et al. Lung Cancer. 2022 Nov;173:43-4.
[15]Yong Fang, et al. 2022 ESMO 1036P.
[16]Lin Wu, et al. 2022 WCLC EP08.02-158.
[17]Zhou Y, et al. Clin Cancer Res. 2022 Oct 13:CCR-22-1903.
[18]Liu J,Guo W,et al. Clin Cancer Res. 2022 Aug 15;28(16):3473-3479.
[19]Xiuli Yang,et al. Journal of Clinical oncology 2023 41:4_suppl, 377-377
[20]Junsheng Wang, et al. Journal of Clinical oncology 2021 39:3_suppl, 181-181.
[21]Chen Jueming, et al. Annals of oncology (2021) 32 (suppl_5): S725-S772. 10.1016/annonc/annonc703.
[22]D. Miller, et al. Annals of Oncology, Volume 30, Supplement 5, 2019, Page v422,ISSN 0923-7534.







